- 1怎么在navicat导入excel文件、csv文件、DBase文件...........?_navicat导入csv文件
- 2高可用的分布式Hadoop大数据平台搭建,超详细,附代码。_四、启动hadoop分布式环境. 写清楚步骤及截图。
- 3【华为OD机试真题 JS语言】432、 密码解密 | 机试真题+思路参考+代码解析(最新C卷抽中)(双代码)_华为c卷密码解密
- 4Linux权限管理_$uid -gt 199
- 5一款挺好看的南瓜影视APP下载页面_dl.keke1.app
- 6Elasticsearch 嵌套类型的深度剖析与实例_elastic search嵌套文档查询
- 7MySQL的触发器、索引和存储引擎
- 8【算法】【二叉树,DFS,哈希集合,分类讨论】力扣1110. 删点成林
- 9python PyQt5窗口运行vue项目记录_pyqt vue
- 10Qt实现简单的显示网页(QtWebkit、QtWebEngine、QAxWidget)_qt 显示网页
病理图像相关内容整理_detecting cancer metastases on gigapixel pathology
赞
踩
乳腺癌论文:
[1] Spanhol, F., Oliveira, L. S., Petitjean, C., Heutte, L., A Dataset for Breast Cancer Histopathological Image Classification, IEEE Transactions on Biomedical Engineering (TBME), 63(7):1455-1462, 2016.
---二区文章------数据集BreaKHis
摘要 - 今天,医学图像分析论文需要坚实的实验来证明所提方法的有效性。然而,通常对研究人员选择的数据进行实验,这些数据可能来自不同的机构,扫描仪和人群。可以使用不同的评估措施,使得难以比较这些方法。在本文中,我们介绍了82名患者获得的7909例乳腺癌组织病理学图像的数据集,现在可从http://web.inf.ufpr.br/vri/breast-cancer-database公开获得。该数据集包括良性和恶性图像。与该数据集相关联的任务是将这些图像自动分类为两类,这对于临床医生来说将是有价值的计算机辅助诊断工具。为了评估这项任务的难度,我们展示了使用最先进的图像分类系统获得的一些初步结果。精度范围从80%到85%,显示出改进的余地。通过向科学界提供此数据集和标准化评估协议,我们希望聚集医学和机器学习领域的研究人员,以推进这一临床应用。
[2] Spanhol, F., Oliveira, L. S., Petitjean, C., and Heutte, L., Breast Cancer Histopathological Image Classification using Convolutional Neural Network, International Joint Conference on Neural Networks (IJCNN 2016), Vancouver, Canada, 2016.
---A类会议,附有Caffe代码------数据集BreaKHis
- Caffe_models.tar.gz – It contains two Caffe model definition files (protobuf model format), a solver file and a trained Caffe model. This model is snapshot of iteration 80,000, considering the strategy #4, fold 1 magnification factor 40x, as described in [1]. Please, edit the files and adjust the paths properly.
- 摘要 - 大多数传统分类系统的性能依赖于适当的数据表示,并且大部分工作都致力于特征工程,这是一个困难且耗时的过程,它使用数据的先前专家领域知识来创建有用的特征。另一方面,深度学习可以从数据中提取和组织判别信息,而不需要域专家设计特征提取器。卷积神经网络(CNN)是一种特殊类型的深度前馈网络,受到研究界和业界的关注,在语音识别,信号处理,对象识别,自然语言处理和转移学习等任务中取得了经验上的成功。在本文中,我们使用深度学习方法对BreaKHis的乳腺癌组织病理学图像进行了一些初步实验,这是一个公共数据集,可在http://web.inf.ufpr.br/vri/breast-cancer-database上找到。我们提出了一种基于图像块的提取的方法,用于训练CNN以及这些块的组合以用于最终分类。该方法旨在允许使用来自BreaKHis的高分辨率组织病理学图像作为现有CNN的输入,避免模型的适应性,这可以导致更复杂且计算成本更高的架构。与之前报告的其他使用手工制作的纹理描述符训练的机器学习模型获得的结果相比,CNN表现更好。最后,我们还使用简单的融合规则研究了不同CNN的组合,实现了识别率的一些改进。
[3] Spanhol, F., Cavalin, P., Oliveira, L. S., Petitjean, C., Heutte, L., Deep Features for Breast Cancer Histopathological Image Classification, 2017 IEEE International Conference on Systems, Man, and Cybernetics (IEEE SMC 2017), Banff, Canada, 2017
---QUALIS b2------数据集BreaKHis
[4]Kraus O Z, Grys B T, Ba J, et al. Automated analysis of high‐content microscopy data with deep learning[J]. Molecular systems biology, 2017, 13(4): 924.
--- 一区 引用61 ----微观 非病理图像 酵母ORF-GFP数据集 ---未找到 网页打不开
用于高内容显微镜数据的定量分析的现有计算流水线依赖于传统的机器学习方法,这些方法在没有实质性调整和训练的情况下无法准确地对多于一个数据集进行分类,需要进行广泛的分析。在这里,我们证明了深度学习在生物图像数据中的应用可以克服与传统机器学习分类器相关的缺陷。使用深度卷积神经网络(DeepLoc)分析酵母细胞图像,我们在蛋白质亚细胞定位的自动分类中显示出优于传统方法的性能。我们还展示了DeepLoc对高度不同图像集进行分类的能力,包括具有异常细胞形态的信息素捕获细胞的图像,以及在不同遗传背景和不同实验室中生成的图像。我们提供了一个开源实现,可以在新的显微镜数据集上更新DeepLoc。本研究强调深度学习是加速分析高内容显微镜数据的重要工具。
[5]Kraus O Z, Ba J L, Frey B J. Classifying and segmenting microscopy images with deep multiple instance learning[J]. Bioinformatics, 2016, 32(12): i52-i59.
---大类二区,小类别一区 引用109
[6]Litjens G, Sánchez C I, Timofeeva N, et al. Deep learning as a tool for increased accuracy and efficiency of histopathological diagnosis[J]. Scientific reports, 2016, 6: 26286.
---三区 引用203 -----无法获取数据集
由于个性化医疗的出现,病理学家面临着组织病理学癌症诊断的工作量和复杂性的显着增加。因此,诊断协议必须同样关注效率和准确性。在本文中,我们引入“深度学习”作为提高组织病理学幻灯片分析的客观性和效率的技术。通过两个例子,活检标本中的前列腺癌鉴定和前哨淋巴结中的乳腺癌转移检测,我们显示了这种新方法减少病理学家工作量的潜力,同时增加了诊断的客观性。我们发现所有含有前列腺癌和乳腺癌微转移和宏观转移的载玻片都可以自动识别,而含有良性和正常组织的载玻片可以排除30-40%,而无需使用任何额外的免疫组化标记或人工干预。我们得出结论,“深度学习”有望提高前列腺癌诊断和乳腺癌分期的疗效。
[7]Wang D, Khosla A, Gargeya R, et al. Deep learning for identifying metastatic breast cancer[J]. arXiv preprint arXiv:1606.05718, 2016.
---arXiv 引用214------ISBI Camelyon16 ISBI Camelyon17 -----已经开始下载
16 第一个训练数据集包括170个淋巴结WSI(100个正常和70个含有转移瘤)和第二个100个WSI(包括60个正常载玻片和40个含有转移的载玻片)。测试集为130个WSI
17年添加了50张label
国际生物医学成像研讨会(ISBI)在评估前哨淋巴结活组织检查的整个幻灯片图像中自动检测转移性乳腺癌的计算系统方面面临巨大挑战。 我们的团队在巨大的挑战中赢得了两场比赛,在整个幻灯片图像分类的任务中获得了0.925的接收器操作曲线(AUC)下的区域,并且对于肿瘤定位任务获得了0.7051的分数。 病理学家独立地审查相同的图像,获得整个幻灯片图像分类AUC为0.966并且肿瘤定位分数为0.733。 将我们的深度学习系统的预测与人类病理学家的诊断相结合,将病理学家的AUC提高到0.995,表示人为错误率降低了大约85%。 这些结果证明了使用深度学习来显着改善病理诊断准确性的能力。
[8]Madabhushi A, Lee G. Image analysis and machine learning in digital pathology: challenges and opportunities[J]. 2016.
---未知 引用127
[9]Janowczyk A, Madabhushi A. Deep learning for digital pathology image analysis: A comprehensive tutorial with selected use cases[J]. Journal of pathology informatics, 2016, 7.
--- 一区 引用205 ---后期再重点看
[10]Van Valen D A, Kudo T, Lane K M, et al. Deep learning automates the quantitative analysis of individual cells in live-cell imaging experiments[J]. PLoS computational biology, 2016, 12(11): e1005177.
---大类二区,小类别一二区 引用87 ---无法获得数据集(大肠杆菌与哺乳动物细胞数据--不相关)
[11]Ciresan D, Giusti A, Gambardella L M, et al. Deep neural networks segment neuronal membranes in electron microscopy images[C]//Advances in neural information processing systems. 2012: 2843-2851.
---a 类会议 引用788
我们解决了神经解剖学的核心问题,即电子显微镜(EM)图像堆栈中描绘的神经元结构的自动分割。这对于有效地映射3D大脑结构和连接是必要的。为了分割生物神经元膜,我们使用特殊类型的深度人工神经网络作为像素分类器。每个像素(膜或非膜)的标签是从以其为中心的方形窗口中的原始像素值预测的。输入层将每个窗口像素映射到神经元。接下来是一系列卷积和最大池化层,它们保留2D信息并随着抽象级别的提高而提取特征。输出层为每个类产生校准概率。分类器通过具有已知地面实况的512×512×30堆栈上的简单梯度下降进行训练,并且由ISBI 2012 EM分段挑战的组织者在相同大小的堆栈(作者未知的基础事实)上进行测试。即使没有特定于问题的后处理,我们的方法在所有三个考虑的度量中都大大超过了竞争技术,即兰德误差,扭曲误差和像素误差。对于像素误差,我们的方法是唯一一个超越第二个人类观察者的方法
[12]Pärnamaa T, Parts L. Accurate classification of protein subcellular localization from high-throughput microscopy images using deep learning[J]. G3: Genes, Genomes, Genetics, 2017, 7(5): 1385-1392.
---3区 引用34
许多单细胞的高通量显微镜检查产生的高维数据远非直接分析。一个重要的问题是自动检测荧光标记的蛋白质所在的细胞区室,这对于有经验的人来说相对简单,但是难以在计算机上自动化。在这里,我们通过绘制数千种酵母蛋白质的数据训练11层神经网络,实现了91%的每细胞定位分类准确度,并且在保持图像上每蛋白质精度达到99%。我们确认低级网络功能对应于基本图像特征,而更深层次分离本地化类。使用该网络作为特征计算器,我们训练标准分类器,在仅观察少量训练样例后,将蛋白质分配给以前看不见的区室。我们的结果是迄今为止最准确的亚细胞定位分类,并证明了深度学习对于高通量显微镜的有用性。
[13]Dong B, Shao L, Da Costa M, et al. Deep learning for automatic cell detection in wide-field microscopy zebrafish images[C]//2015 IEEE 12th International Symposium on Biomedical Imaging (ISBI). IEEE, 2015: 772-776.
---QUALIS b1 引用32 -----未下载
斑马鱼已成为生物医学研究的流行实验模型生物。 在本文中,提出了一个独特的框架,用于自动检测通过宽视野显微镜记录的幼虫斑马鱼脑z-堆叠图像中含酪氨酸羟化酶(TH标记)的细胞。 在该框架中,训练监督的最大池汇集卷积神经网络(CNN)以检测由支持向量机(SVM)分类器预先选择的区域中的单元像素。 结果表明,所提出的深度学习方法优于手工制作技术,并证明了其在宽视场显微镜z-stack斑马鱼图像中自动细胞检测的潜力。
该数据集包含通过宽视场显微镜在20X放大率下扫描的35个堆叠。 选择25个这样的堆栈用于离线训练,10个堆栈用于测试。 总共有大约1000个TH标记的细胞。 每个堆栈的体积(以体素为单位)是1024 * 1344 * z,并且不同的堆栈具有不同的z值。 空间分辨率为3μm,轴向分辨率为1.5μm。 物镜的放大倍数为20倍,数值孔径(NA)为0.7。 我们将此数据集公开提供给其他研究人员,以开发和评估他们的TH标记细胞检测或细胞计数算法。 对于该数据集中的每个堆栈,专业观察者使用“Point Picker”[19]标记所有TH标记单元格的中心作为基础事实,这是开源JAVA程序“ImageJ”[11]的插件。
1The link for downloading the dataset will be released in the final version of our paper:http://www.cistib.com/cistib_shf/index. php/translation/downloads
---[14]Xu Y, Mo T, Feng Q, et al. Deep learning of feature representation with multiple instance learning for medical image analysis[C]//2014 IEEE international conference on acoustics, speech and signal processing (ICASSP). IEEE, 2014: 1626-1630.
---CCF b 引用156 ---没有数据集 浙江清华 --没找作者要
本文研究了用最少的手工注释完成高级任务的有效性和医学图像的良好特征表示。在医学图像分析中,像细胞这样的物体具有显着的临床特征。先前开发的诸如SIFT和HARR的特征不能全面地表示这样的对象。因此,特征表示尤其重要。在本文中,我们研究通过深度学习(DNN)自动提取特征表示。此外,对象的详细注释通常是模糊且具有挑战性的任务。我们在具有深度学习功能的分类培训中使用多实例学习(MIL)框架。从我们的工作中可以得出几个有趣的结论:(1)自动特征学习优于手动特征; (2)无监督方法可以达到接近完全监督方法的性能(93.56%)与(94.52%); (3)粗略标签的MIL性能(96.30%)超过监督深度学习特征中的精细标签(95.40%)的监督性能。
高分辨率组织病理学图像用于构建我们的数据集。所有图像均选自132名患者的组织病理学图像。由于单个机器的计算能力,每个图像被设置为10000x10000像素。这是MIL提到的一个包。我们采样200x200像素的补丁,而重叠步长是100像素,因此我们在一个图像中获得了9801个补丁,每个补丁都是一个实例。详细的数据集如下(见表1):完全监督的数据集:首先,我们选择了30个癌症图像,我们对细胞进行了手动分割。将完全封闭在标记的癌症区域内的9000个贴剂用作阳性实例。从30个非癌症图像中,我们随机抽取9000个补丁作为阴性实例。从上述数据中随机选择训练集和测试集,其中包含4500个正实例和4500个负实例。训练集不仅用于训练完全监督学习算法,如SVM和DNN,而且也是弱监督学习的评估数据集。图2显示了几个补丁的例子。弱监督数据集:30个没有手动分割的正面图像和83个负面图像被用作包装袋,每个图像包含9801个贴片,因此我们有113个标记袋和超过100万个未标记的实例来构建MIL模型。注释:完全监督(癌症区域)和弱监督(袋标签)的注释均由两位病理学家独立标记。当出现分歧时,第三位高级病理学家将与他们讨论确定基本事实。
[15]Quan T M, Hildebrand D G C, Jeong W K. Fusionnet: A deep fully residual convolutional neural network for image segmentation in connectomics[J]. arXiv preprint arXiv:1612.05360, 2016.
---aRxiv 引用45
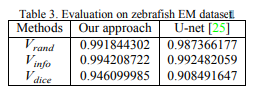
---ISBI EM 斑马鱼 --已经获取0.991844302 0.994208722 0.946099985
[16]Bayramoglu N, Kannala J, Heikkilä J. Deep learning for magnification independent breast cancer histopathology image classification[C]//2016 23rd International conference on pattern recognition (ICPR). IEEE, 2016: 2440-2445.
--- CCF c C4ORE b QUALIS a1 引用43
-----BreakHis 已经获取
[17]Min S, Lee B, Yoon S. Deep learning in bioinformatics[J]. Briefings in bioinformatics, 2017, 18(5): 851-869.
---一区 引用286 貌似不是很相关
[18]Akram S U, Kannala J, Eklund L, et al. Cell segmentation proposal network for microscopy image analysis[M]//Deep Learning and Data Labeling for Medical Applications. Springer, Cham, 2016: 21-29.
---书籍
[19]Liu Y, Gadepalli K, Norouzi M, et al. Detecting cancer metastases on gigapixel pathology images[J]. arXiv preprint arXiv:1703.02442, 2017.
---arXiv 引用129 92.4%

[20]Rakhlin A, Shvets A, Iglovikov V, et al. Deep convolutional neural networks for breast cancer histology image analysis[C]//International Conference Image Analysis and Recognition. Springer, Cham, 2018: 737-744.
---core c 引用22 主要是有代码 https://github.com/alexander-rakhlin/ICIAR2018
[21]Bychkov D, Linder N, Turkki R, et al. Deep learning based tissue analysis predicts outcome in colorectal cancer[J]. Scientific reports, 2018, 8(1): 3395.
---三区 引用32
[22]iang Z, Powell A, Ersoy I, et al. CNN-based image analysis for malaria diagnosis[C]//2016 IEEE International Conference on Bioinformatics and Biomedicine (BIBM). IEEE, 2016: 493-496.
---b类会议 引用22 疟疾 相关性不大 文章简单
[23]Chen H, Qi X J, Cheng J Z, et al. Deep contextual networks for neuronal structure segmentation[C]//Thirtieth AAAI conference on artificial intelligence. 2016.
---a类会议 引用71 ISBI EM
[24]Rivenson Y, Göröcs Z, Günaydin H, et al. Deep learning microscopy[J]. Optica, 2017, 4(11): 1437-1443.
---一区 引用104 图像增强
[25]Xie W, Noble J A, Zisserman A. Microscopy cell counting and detection with fully convolutional regression networks[J]. Computer methods in biomechanics and biomedical engineering: Imaging & Visualization, 2018, 6(3): 283-292.
---三区 引用113
[26]Fakhry A, Zeng T, Ji S. Residual deconvolutional networks for brain electron microscopy image segmentation[J]. IEEE transactions on medical imaging, 2017, 36(2): 447-456.
---二区 引用36
[27]Araújo T, Aresta G, Castro E, et al. Classification of breast cancer histology images using convolutional neural networks[J]. PloS one, 2017, 12(6): e0177544.
---三区 引用112
[28]Komura D, Ishikawa S. Machine learning methods for histopathological image analysis[J]. Computational and structural biotechnology journal, 2018, 16: 34-42.
---二区 引用32 可用来获取数据集
[29]Aubreville M, Knipfer C, Oetter N, et al. Automatic classification of cancerous tissue in laserendomicroscopy images of the oral cavity using deep learning[J]. Scientific reports, 2017, 7(1): 11979.
---三区 引用32 口腔
[30]Teramoto A, Tsukamoto T, Kiriyama Y, et al. Automated classification of lung cancer types from cytological images using deep convolutional neural networks[J]. BioMed research international, 2017, 2017.
---三区 引用16 肺癌
[31]Drozdzal M, Chartrand G, Vorontsov E, et al. Learning normalized inputs for iterative estimation in medical image segmentation[J]. Medical image analysis, 2018, 44: 1-13.
---二区 引用46 ---无法查看文章
[32]Pan X, Li L, Yang H, et al. Accurate segmentation of nuclei in pathological images via sparse reconstruction and deep convolutional networks[J]. Neurocomputing, 2017, 229: 88-99.
---二区 引用27---无法查看文章
[33]Khosravi P, Kazemi E, Imielinski M, et al. Deep convolutional neural networks enable discrimination of heterogeneous digital pathology images[J]. EBioMedicine, 2018, 27: 317-328.
---二区 引用30
[34]Nguyen T, Bui V, Lam V, et al. Automatic phase aberration compensation for digital holographic microscopy based on deep learning background detection[J]. Optics express, 2017, 25(13): 15043-15057.
---二区 引用38
[35]Raza S E A, Cheung L, Epstein D, et al. Mimo-net: A multi-input multi-output convolutional neural network for cell segmentation in fluorescence microscopy images[C]//2017 IEEE 14th International Symposium on Biomedical Imaging (ISBI 2017). IEEE, 2017: 337-340.
---b1 引用14
[36]Chen H, Qi X, Yu L, et al. DCAN: Deep contour-aware networks for object instance segmentation from histology images[J]. Medical image analysis, 2017, 36: 135-146.
---二区 引用81 2015 MICCAI Gland Segmentation Challenge and 2015 MICCAI Nuclei Segmentation Challenge
数据集:
乳腺癌数据集:82名患者的7909张图像,放大因子为40、100、200、400。2480良性,5429个恶性,大小为700 X 460,3通道RGB图像,8-bit depth ----------已申请
相关操作:


