- 1贪吃蛇大作战双人版完整代码
- 2基于微信小程序的自助点餐系统的详细设计和实现(源码+lw+部署文档+讲解等)_自助点菜系统设计
- 3DataStage:DataStage Job优化指导原则(不定时更新中)_datastage无法改名job
- 4【Java】ios应用开发证书申请流程_ios开发者证书申请教程
- 5USB3.0接口——(2)数据结构_usb的trb
- 6[深度学习-NPL]ELMO、BERT、GPT学习与总结_npl gpt
- 7【分库分表】sharding-jdbc(shardingsphere)—分片策略_sharding jdbc 分片策略
- 8python 机器学习_Python机器学习-知识点汇总
- 9排序算法的原理、实现、优缺点_排序精选类算法的风险
- 10通俗易懂的自定义view详解
python处理让3Dslicer标注更加细腻_3d slicer 标注
赞
踩
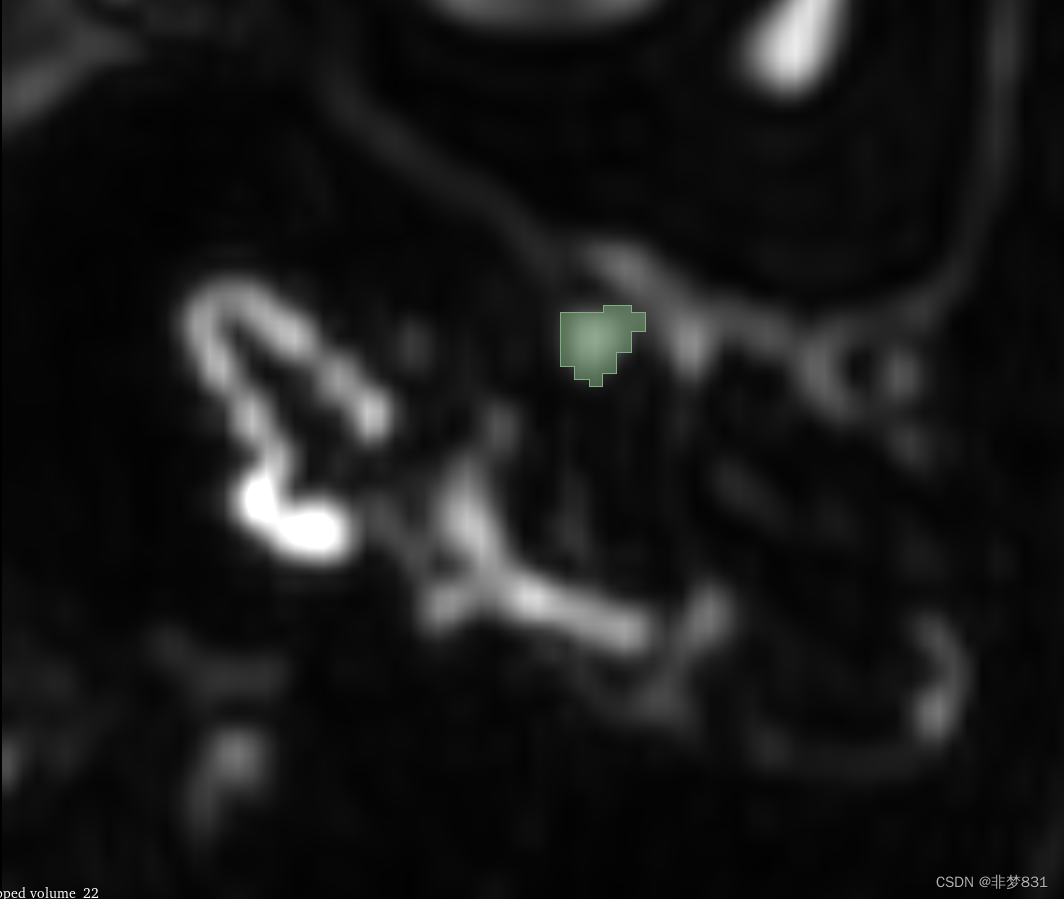
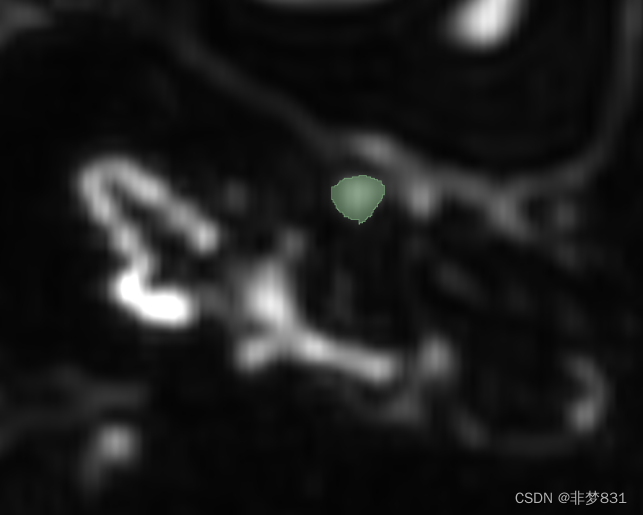
针对小目标,在做医学图像分割的时候,标注软件对于小目标的标注是基于像素点的,由于是小目标,所以标注出来的误差会非常大,这会导致我们分割出来的图像边缘锐化会非常严重,而且分割不准确,在我们的接下来的处理中,可以让你的.nii格式的医学图像对于3D分割的结果精确度,更上一层楼.
一.我们的优势.
1.我们通过一种超分算法网络(srcnn)使.nii文件在三个方向轴上的图片分辨率增加,这种算法不会让图像信息发生较大的变化.
2.在三个轴上图片信息可以通过算法增加,针对切片过厚,或者轴上图片数量过少.
3.我们是将每个轴上的图片数据提取出来,使用超分算法,然后生成新的轴数据.产生新的.nii文件.
二.所用到的模型算法原理
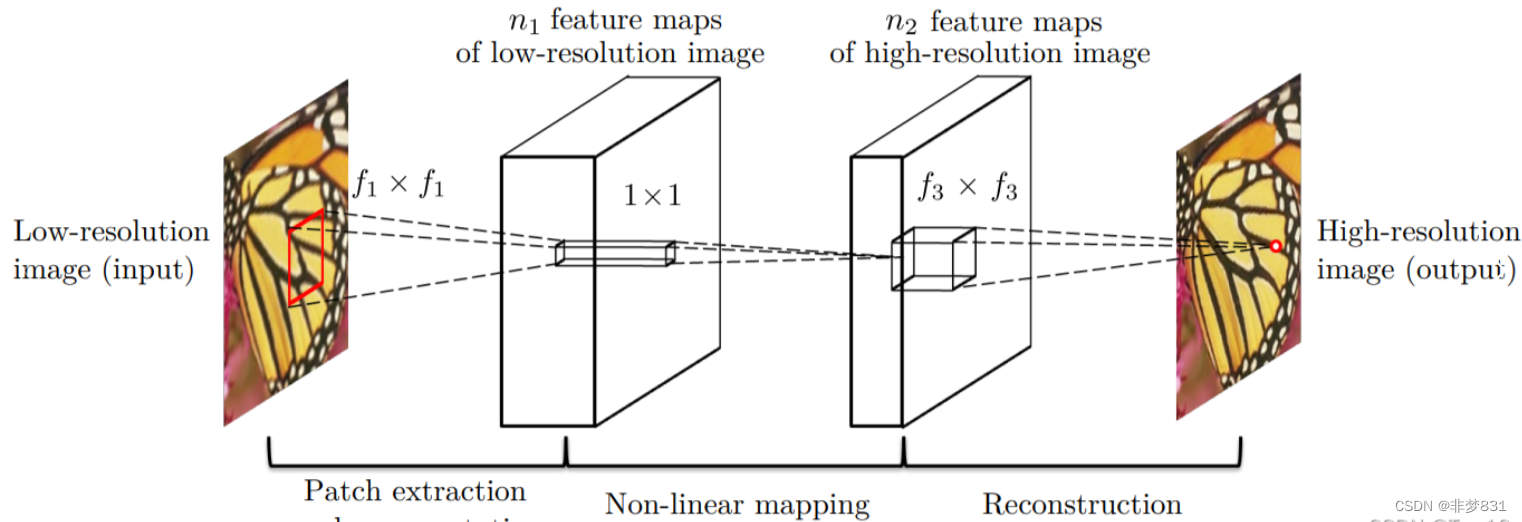
SRCNN【1】是end-to-end(端到端)的超分算法,所以在实际应用中不需要任何人工干预或者多阶段的计算,
实际上,SRCNN需要一个预处理过程:将输入的低分辨率图像进行bicubic插值(双三次插值)。
符号规定如下:
(a)插值后的图像依旧称为“低分辨率图像”,并用Y表示;
(b)将ground-truth(真实的高分辨率图像)用X表示;
(c)将网络记为映射函数F(·);
SRCNN网络包含三个模块:Patch extraction and representation(块析出与表示)、Non-linear mapping(非线性映射)、Reconstruction(重构)。这三个模块对应三个卷积操作,接下来分别进行分析:
(1)Patch extraction and representation
块析出和表示的目的是通过输入图像Y获得一系列特征图:
![]()
其中W1和B1表示滤波器(卷积核)的权重和偏置,max操作对应ReLU激活函数,所以这实际上就是卷积+激活操作。
(2)Non-linear mapping
非线性映射对应的也是“卷积+激活”操作:
![]()
其中W2和B2依旧表示滤波器的权重和偏置(这里作者的意思是可以添加更多的层,但是会增加网络的计算开销)。
(3)Reconstruction
重构过程同样是卷积操作,但是这里没有激活函数了:
![]()
2、损失函数
不同于今天的深度卷积神经网络包含大量的参数,如VGG系列、ResNet系列和DenseNet系列,SRCNN只有6个需要学习的参数{W1,B1,W2,B2,W3,B3}。损失的计算也仅仅需要网络的输出F(Y)与真实高分图像X,损失函数选择MSE损失.
具体由于篇幅原因可以上gitup或其他途径了解srcnn.
代码如下:
需要的权重文件在我的百度
网盘链接:https://pan.baidu.com/s/1Fk1BwQP-yU1XkS4DodcLLg?pwd=gwdz
提取码:gwdz
文中的缩放因子需要电脑的运行内存,如果爆内存请改小.
import numpy as np
import nibabel as nib
import torch
from torch import nn
from torchvision.transforms import ToTensor
from skimage.transform import resize
import os
from scipy.ndimage import zoom
os.environ['KMP_DUPLICATE_LIB_OK'] = 'True'
# 自定义SRCNN模型
class SRCNN(nn.Module):
def __init__(self):
super(SRCNN, self).__init__()
self.conv1 = nn.Conv2d(1, 64, kernel_size=9, padding=4)
self.conv2 = nn.Conv2d(64, 32, kernel_size=5, padding=2)
self.conv3 = nn.Conv2d(32, 1, kernel_size=5, padding=2)
def forward(self, x):
x = nn.functional.relu(self.conv1(x))
x = nn.functional.relu(self.conv2(x))
x = self.conv3(x)
return x
# 加载SRCNN模型权重
model_path = r"请输入你的模型的地址"
srcnn_model = SRCNN()
srcnn_model.load_state_dict(torch.load(model_path))
srcnn_model = srcnn_model.float() # 将模型的权重类型转换为 Float
srcnn_model.eval()
# 设置输入和输出文件夹路径
input_folder = r"你的要做的.nii文件夹"
output_folder = r"输出结果的保存地址"
input_nii_files = [file for file in os.listdir(input_folder) if file.endswith('.nii.gz')]
# 获取输入文件夹内所有.nii文件的路径
input_nii_files = [file for file in os.listdir(input_folder) if file.endswith('.nii.gz')]
# 创建输出文件夹(如果不存在)
if not os.path.exists(output_folder):
os.makedirs(output_folder)
# 定义缩放因子
scale_factor = 4 # 请根据您的需求设置适当的值
# 循环处理每个输入文件
for nii_filename in input_nii_files:
nii_path = os.path.join(input_folder, nii_filename)
nii_data = nib.load(nii_path)
nii_affine = nii_data.affine
# 获取NIfTI文件的头信息
nii_header = nii_data.header
# 获取图像的维度信息
image_shape = nii_header.get_data_shape()
# 获取处理后的切片
processed_slices = nii_data.get_fdata()
# 初始化新的3D图像数组
new_shape = [int(dim * scale_factor) for dim in image_shape]
new_data = np.empty(new_shape)
# 处理每个轴
axis_output_data_list = []
for axis in range(len(image_shape)):
print("Processing axis:", axis)
axis_slices = np.take(processed_slices, indices=range(processed_slices.shape[axis]), axis=axis)
print("Axis slices shape:", axis_slices.shape)
# 超分辨率重建代码
output_slices = []
for slice_idx in range(axis_slices.shape[0]):
input_slice = axis_slices[slice_idx, ...]
# 调整输入切片的尺寸,使其符合模型的输入尺寸
input_slice_resized = resize(input_slice, output_shape=(
input_slice.shape[0] * scale_factor, input_slice.shape[1] * scale_factor), mode='reflect',
anti_aliasing=True)
input_tensor = ToTensor()(input_slice_resized).float().unsqueeze(0) # 将输入数据类型转换为 Float
# 使用SRCNN模型进行超分辨率重建
with torch.no_grad():
output_tensor = srcnn_model(input_tensor).squeeze().cpu().numpy()
output_slices.append(output_tensor)
# 将处理后的切片重新组合成轴数据
axis_output_data = np.stack(output_slices, axis=axis)
axis_output_data_list.append(axis_output_data)
# 对每个轴的数据进行缩放并调整形状
zoomed_data_list = []
for axis in range(len(image_shape)):
zoomed_axis_data = zoom(axis_output_data_list[axis], scale_factor, mode='reflect')
zoomed_data_list.append(zoomed_axis_data)
# 将所有轴的数据都扩大到目标形状
target_shape = tuple(new_shape)
for axis in range(len(image_shape)):
zoomed_data_list[axis] = resize(zoomed_data_list[axis], target_shape, mode='reflect')
# 创建新的空数组,其形状与目标形状相同
combined_data = np.empty(target_shape)
# 将每个轴的数据放入新数组的相应位置
for axis in range(len(image_shape)):
slices_indices = [slice(None)] * len(image_shape)
slices_indices[axis] = slice(None)
slices_indices = tuple(slices_indices)
combined_data[slices_indices] = zoomed_data_list[axis]
# 保存输出NIfTI图像
output_nii = nib.Nifti1Image(combined_data, nii_affine)
output_nii_filename = nii_filename.replace('.nii.gz', '_output.nii.gz')
output_nii_path = os.path.join(output_folder, output_nii_filename)
nib.save(output_nii, output_nii_path)
print("Output NIfTI image with enhanced resolution saved at:", output_nii_path)


